1、百度搜索:BioEdit下载
2、安装之后,打开软件,显示以下画面:
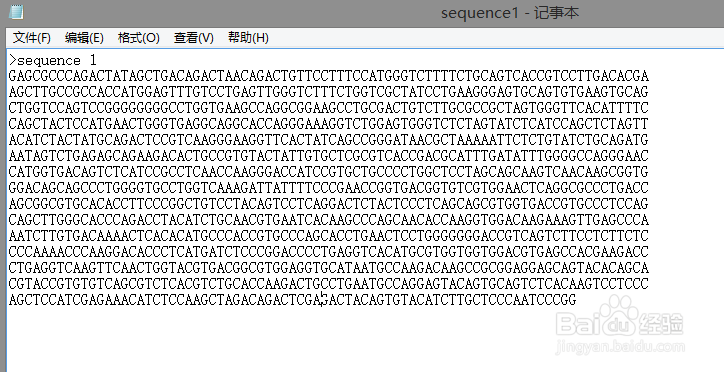
3、将序列粘到文本文档中,如:“sequence1”,注意用fasta格式,见下图:
4、依次点击BioEdit软件的"file>open>"将“文本文档 sequence1”打开:
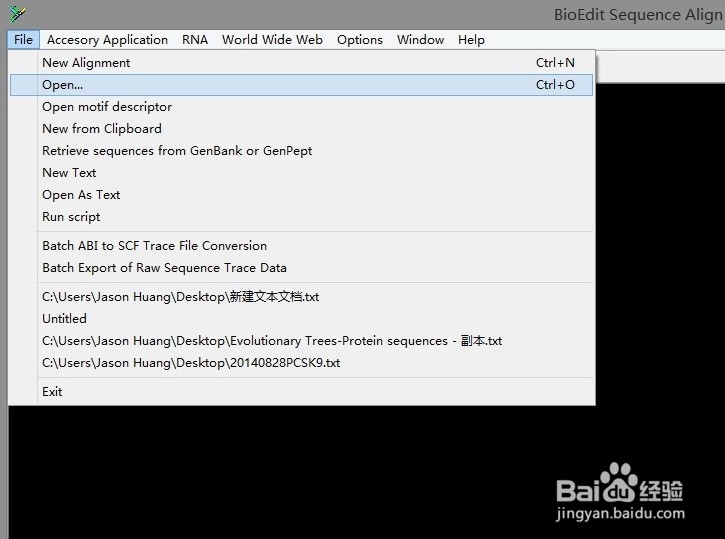
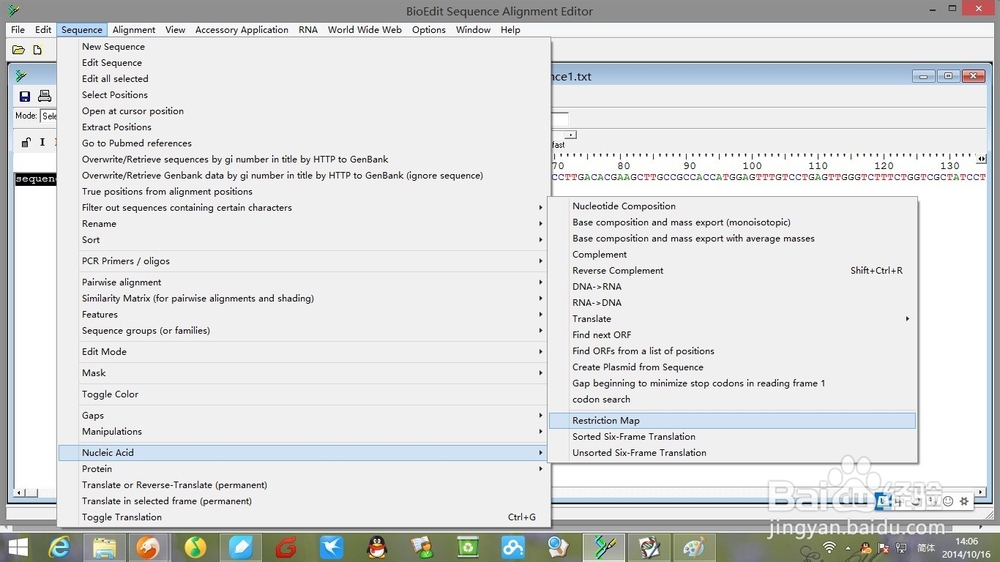
5、选中“sequence1”后,选中标题栏中“sequence”>"Nucleic Acid">"Restriction Map"
6、在新的界面中选择“Generate Map”
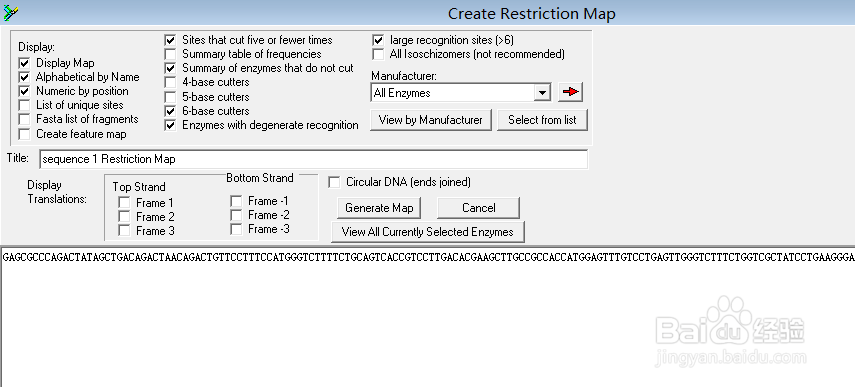
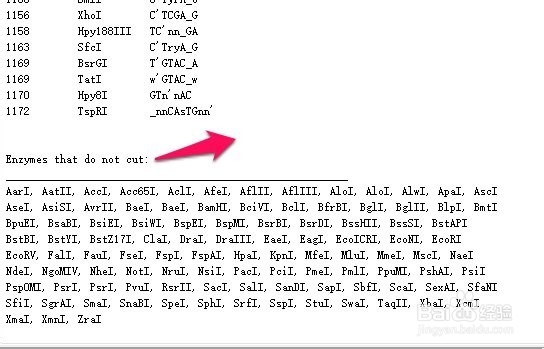
7、在出现的界面中,即是分析得到的酶切位点图,可以显示每个酶切位点在序列中的位置,拖滚动条到最下面,可以看到酶不能切割的酶切位点。